- 首页
- 资讯
Sci. Adv.:清华刘凯、张洪杰团队开发活细胞内DNA大数据存储与定点修改系统
来源:清华大学新闻网 2022-08-11
导读:近日,清华大学化学系刘凯副教授、张洪杰院士团队在《科学·进展》(Science Advances)期刊发表了题为“活细胞内高靶专一性和高鲁棒性的数字信息分子级处理系统”(In vivo processing of digital information molecularly with targeted specificity and robust reliability)的研究论文。文章展示,在大肠杆菌活细胞内,文字、密码表、图片等信息可以被长期稳定的存储、修改和复制,并开发了一种双质粒编辑系通用于准确处理微生物载体中的数字信息。
DNA信息存储技术是近年来信息技术和生物技术等学科交叉的新领域,旨在解决大数据存储对发展新介质的迫切需求。DNA作为信息存储介质,具有极强的稳定性、高存储密度和低维护成本等优点。数字信息可以通过算法编码和DNA合成转变为DNA碱基序列,实现数据的分子级超高密度存储。目前,DNA数据存储技术有两种模式,即“体外硬盘模式”和“体内CD模式”。基于细胞基因组自我复制的机制和特点,“体内模式”相对于“体外模式”的优点主要是信息的可控复制、低成本且可靠性强,使得这一类DNA存储模式可用于快速、低成本的数据拷贝和传播。然而,DNA信息存储“体内CD模式”的难点在于如何将“冷存储”改进为“热存储”——使数字信息不仅能在DNA序列中被灵活地“写入”“保存”“读取”,还可以实现精确的信息“修改”。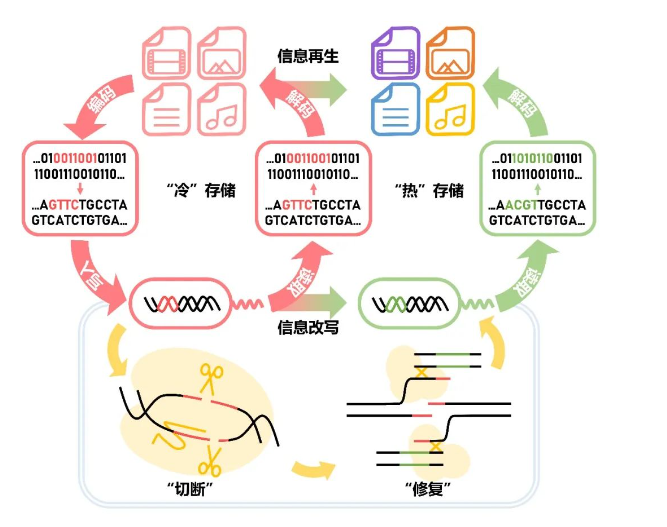
研究人员通过利用CRISPR/Cas基因编辑技术,在活细胞中构建了集存储与改写功能于一体的双质粒信息存储体系,与已有的DNA信息存储方式相比,在降低写入信息冗余度、提高活细胞信息存储能力、简化信息读取流程、提升信息保存安全性上都有显著提升。该研究充分探索了DNA序列的编码能力,不需要任何寻址索引和备份序列,并兼容多种编码算法,最高的编码效率可达每个核苷酸4.0位(4.0bits/nucleotide)。值得注意的是,该研究设计的信息存储与定点修改技术,不仅可以利用活细胞对外源数字信息实现高密度存储和稳定拷贝,还能利用活细胞内的蛋白质工具实现灵活的信息处理。利用预设并优化的CRISPR-Cas12a体系导向crRNA结合序列,可以实现与现有基因编辑相媲美的编辑成功率,并完成在分子水平精准靶向多种类型复杂信息的信息修改处理目标。另外,创新性引入荧光蛋白作为“报告器”进一步使信息改写可视化,极大地提高了改写信息的读取鲁棒性,使分子水平信息存储和修改的状态直观可见。这一研究解决了DNA作为存储介质无法对大数据信息进行精准改写的难点,克服了DNA基质只能作为冷数据存储的弊端,提升了DNA作为信息热存储介质的能力。清华大学化学系为第一完成单位,化学系2020级博士研究生刘杨奕为论文第一作者,清华大学化学系副教授刘凯为通讯作者。清华大学化学系张洪杰院士和上海交通大学樊春海院士给予了大力指导,研究得到了国家自然科学基金、科技部重点研发计划、清华大学春风基金的支持。论文链接:https://www.science.org/doi/10.1126/sciadv.abo7415 参考资料:https://www.tsinghua.edu.cn/info/1175/97183.htm
声明:化学加刊发或者转载此文只是出于传递、分享更多信息之目的,并不意味认同其观点或证实其描述。若有来源标注错误或侵犯了您的合法权益,请作者持权属证明与本网联系,我们将及时更正、删除,谢谢。 电话:18676881059,邮箱:gongjian@huaxuejia.cn


